【
儀表網 研發快訊】近日,南方科技大學生物醫學工程系副教授李依明課題組在高效單分子定位方法研究領域取得重要進展,相關成果以“Scalable and lightweight deep learning for efficient high accuracy single-molecule localization microscopy”為題發表于國際權威期刊Nature Communications。
單分子定位
顯微鏡(single-molecule localization microscopy,SMLM)是目前主流三大超分辨顯微鏡之一,因其兼具超高的空間分辨率和分子特異性,在眾多顯微成像技術中擁有獨特的優勢。近年來,深度學習方法的引入顯著提升了SMLM在低信噪比與高密度樣本下的定位精度。目前,以高內涵篩選(high-content screening)為目標的高通量超分辨成像技術已成為SMLM發展的主要方向。現有深度學習網絡因其模型復雜度高,在高通量成像場景中普遍存在處理延時長、資源消耗大等問題,嚴重制約了其在實際應用中的部署。雖然已有一些模型壓縮技術可降低網絡復雜度,但往往以犧牲定位精度為代價,難以滿足SMLM對高精度定位的需求。
為解決上述問題,研究團隊提出了一種名為LiteLoc的基于輕量化深度學習網絡的可擴展分析框架。該框架結合了由粗、細特征提取器構成的輕量化深度學習網絡與一套可擴展的競爭式并行數據分析策略。在8張RTX 4090顯卡上,LiteLoc實現了超過560 MB/s的吞吐量,能夠實時分析由現代sCMOS相機采集的高通量超分辨成像數據。本研究為基于深度學習的SMLM設立了一個兼顧定位精度與計算效率的新基準,為生命科學領域的高效、可擴展成像工作流程提供了高效的解決方案。
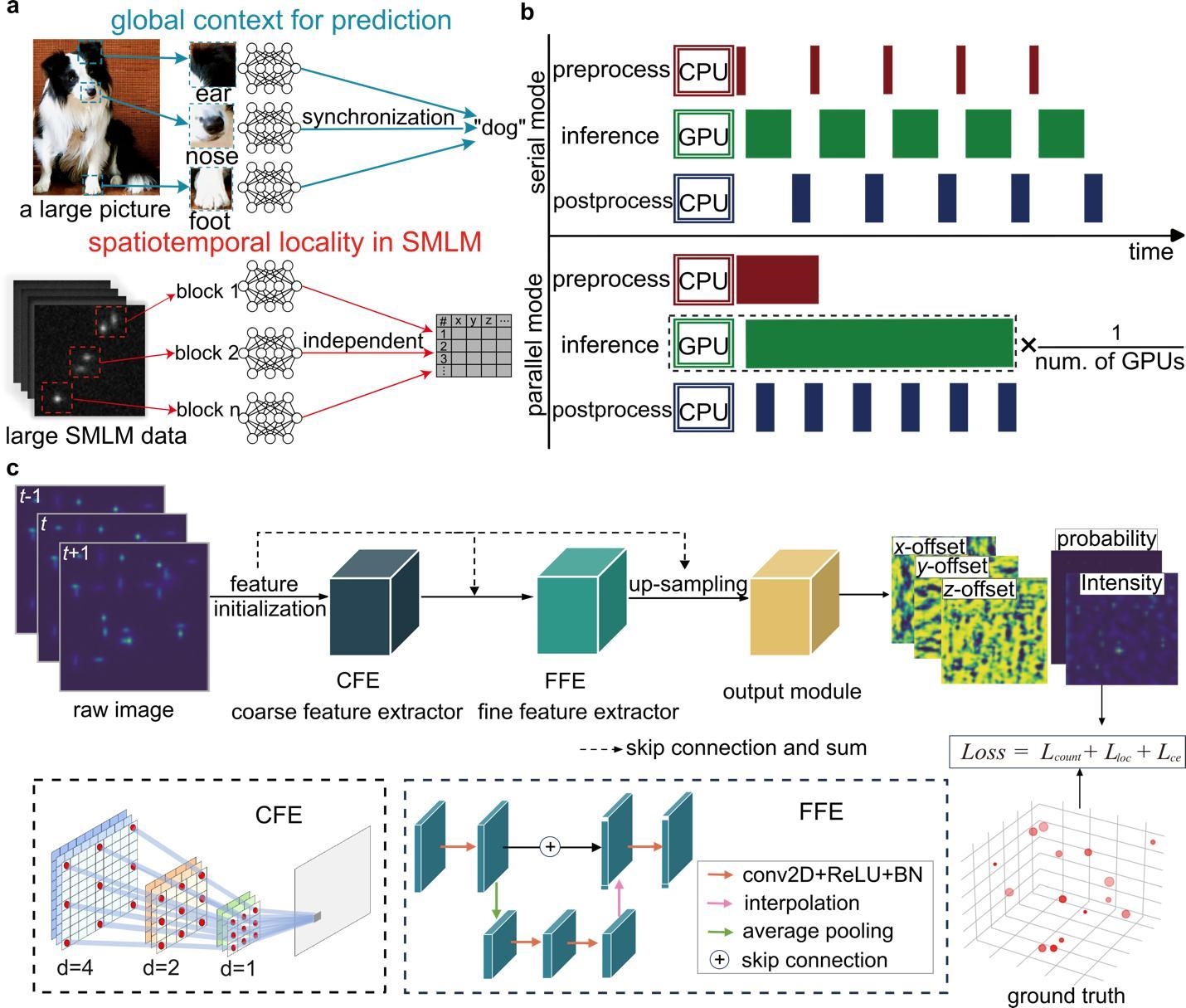
通常傳統圖像中的目標特征分布在整張圖像的不同區域,相比之下,單分子定位數據可被分解為大量時空分離的模塊,每個模塊僅包含局部熒光分子的相關信息,彼此之間相互獨立(如圖1a所示)。然而,現有基于深度學習的單分子數據分析軟件多采用串行處理模式,數據分析流程中CPU和GPU資源未被充分利用。LiteLoc提出的并行分析框架可以最大限度地利用硬件計算資源,將整個數據分析流程模塊化為數據讀取/預處理、網絡推理、后處理/寫入三個階段,并通過優化不同階段的并行處理機制,允許多張顯卡以競爭方式同時讀取與分析處理數據。最終數據分析總耗時與子進程推理幾乎一致(如圖1b所示)。
圖1 LiteLoc的可擴展并行框架和輕量化深度學習網絡示意圖
傳統卷積神經網絡通常通過堆疊多個卷積層來提取多尺度特征。為實現更高效率,空洞卷積因其在維持參數與計算量不變的同時能擴展感受野而受到關注。LiteLoc的輕量化深度學習網絡結構由兩部分組成:一是由空洞系數遞增的空洞卷積組構成的粗特征提取器,二是由簡化版U-Net構成的細特征提取器(如圖1c所示)。該結構通過增大感受野、增強特征復用效率與融合多尺度信息,有效地將模型復雜度降低了一個數量級(如圖2a所示)。與此同時,LiteLoc在定位精度上與當前最優算法DECODE相當甚至更優(如圖2c-f所示)。結合GPU集群并行分析框架,LiteLoc實現了567.6 MB/s的總分析速度,超過常規SATA SSD的讀取上限(500 MB/s)。
圖 2 LiteLoc的分析速度及定位表現
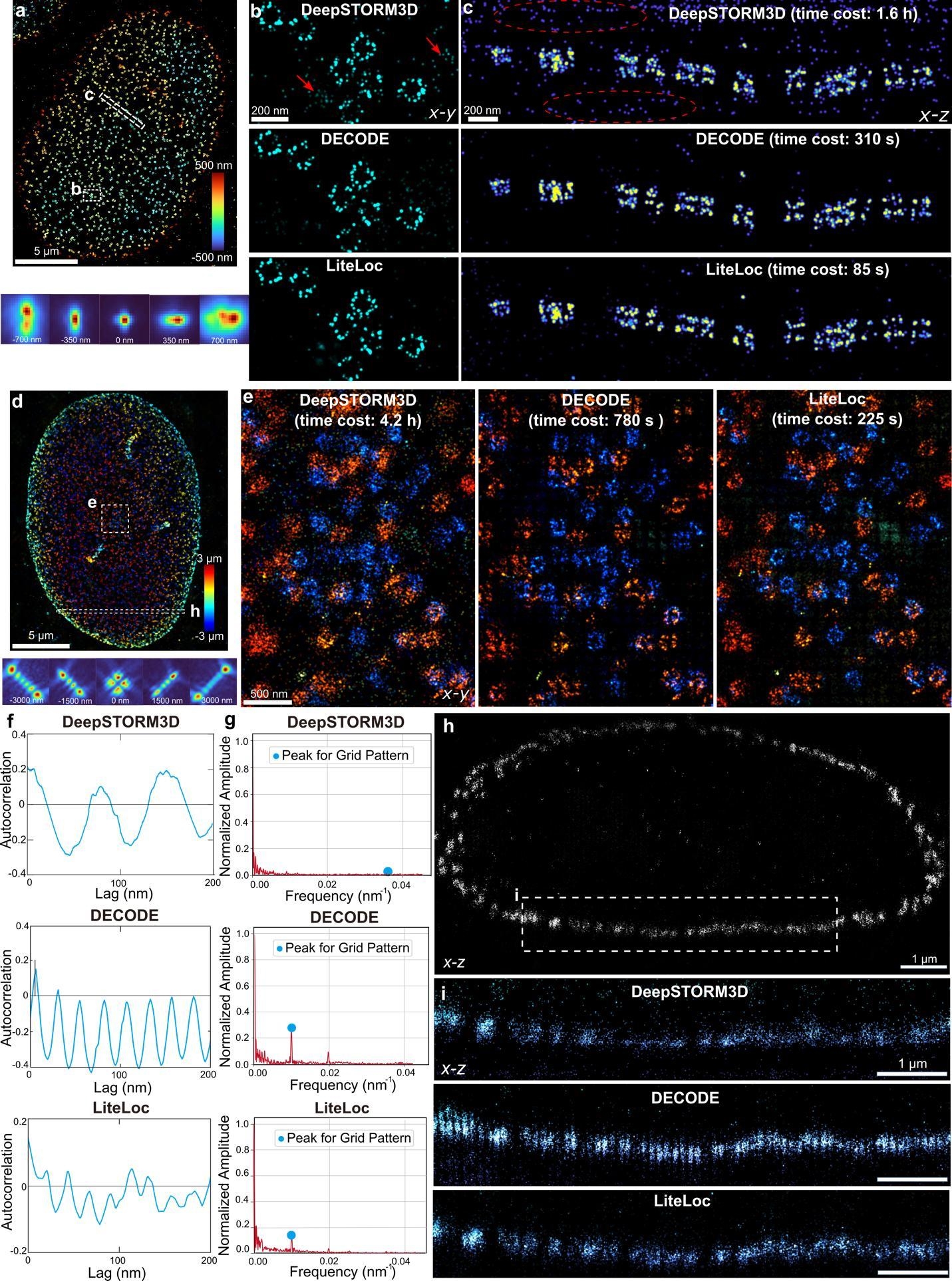
為驗證LiteLoc的定位性能,研究團隊分別對基于散光點擴散函數(point spread function,PSF)和6 µm DMO-Tetrapod PSF對
標準結構U2OS細胞中的核孔蛋白Nup96成像數據進行分析和重建(如圖3所示)。已有研究指出,Nup96雙層環結構平均直徑約為107 nm,雙層環間的平均距離約為50 nm。在使用散光PSF的成像實驗中,LiteLoc與主流定位算法DeepSTORM3D、DECODE均可在x-y平面中重構出核孔的環狀結構(圖3b),并在x-z平面準確解析出靠近蓋玻片的雙層環結構(圖3c)。在6 µm DMO-Tetrapod PSF實驗中,三種算法均可大致重構細胞上下表面的核孔蛋白分布(圖3e)。
然而,由于單個分子的光子分布范圍更大、原始顯微圖像信噪比較低,DeepSTORM3D預測的定位點較為分散,數量也相對較少(圖3d);而DECODE重構的超分辨圖像中出現了明顯的網格狀偽影。相比之下,LiteLoc重構圖像未出現此類偽影(圖3i),顯示出LiteLoc具有更高的定位準確度。在分析速度方面,在相同硬件條件下,LiteLoc的分析耗時僅為DECODE的28.8%、DeepSTORM3D的1.5%,顯著提升了單分子數據的處理效率。
圖3 LiteLoc在基于散光PSF和6 µm DMO-Tetrapod PSF對核孔蛋白Nup96的重構結果
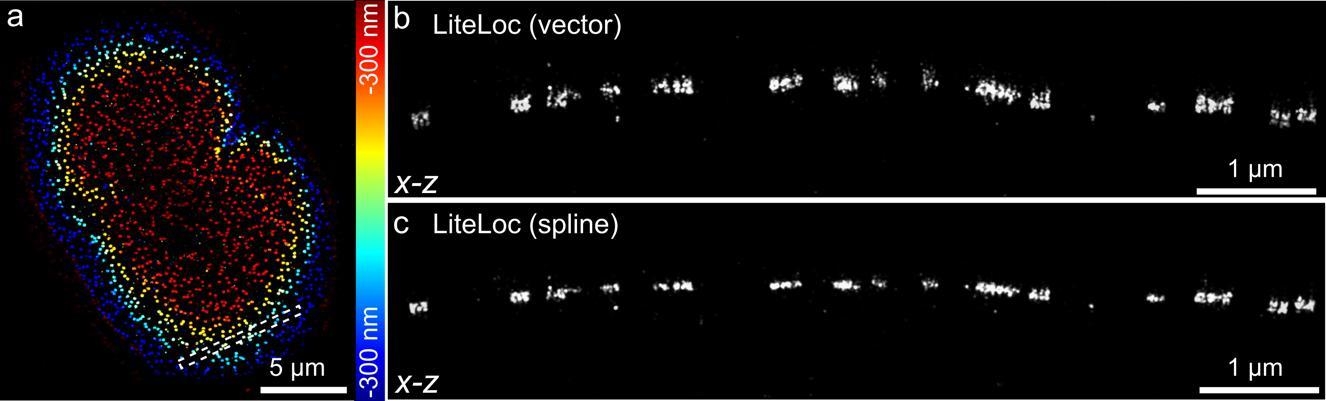
常規的深度學習SMLM軟件支持的PSF建模方式有限,而在實際成像中研究者往往需根據具體成像需求選擇不同建模方式。樣條插值PSF較為簡單,適用于空間不變PSF建模;而矢量PSF考慮多種光學參數,如波長、折射率與數值孔徑,更適合用于包含深度與視場依賴像差的場景。LiteLoc同時支持兩種PSF建模方式用于訓練。圖4展示了結合原位PSF估計與矢量PSF模型訓練的LiteLoc的重構結果,同時將樣條插值PSF訓練的LiteLoc結果作為對比。可以看到基于原位矢量PSF訓練的LiteLoc預測結果與實際NPC結構更加吻合,表明其更適合用于補償深度依賴的像差。
圖4 分別基于矢量PSF和C-spline插值PSF訓練的LiteLoc重建結果
綜上所述,研究團隊提出了一套面向高通量SMLM的輕量化深度學習可擴展并行分析框架。該方法在保證高定位精度的前提下,將網絡推理速度提升三倍以上,顯著緩解了PSF復雜性、結構化數據偽影和成像條件多樣性帶來的挑戰,適用于不同生物樣本的超分辨成像任務。其在8張RTX 4090 GPU上的總處理速度超過560 MB/s,具備實時處理能力,未來有望應用于閉環成像系統與在線質量控制。此外,LiteLoc對計算資源的需求較低,便于集成至標準SMLM分析流程,并與聚類、追蹤或結構重建等下游模塊結合,實現高效一體化的超分辨數據處理流程。
南科大2025屆碩士生費悅、博士后傅爽以及石偉為論文共同第一作者,李依明為該論文通訊作者,南方科技大學為第一通訊單位。該項目得到了國家重點研發計劃、深圳市醫學研究專項資金、國家自然科學基金委員會和南方科技大學校長卓越博士后資金等科研項目的支持。





所有評論僅代表網友意見,與本站立場無關。